การค้นพบโครงสร้างโปรตีนจากแบคทีเรียที่ทำให้เกิดโรคมะเร็งกระเพาะอาหาร
เขียนโดย อร่าม นราพล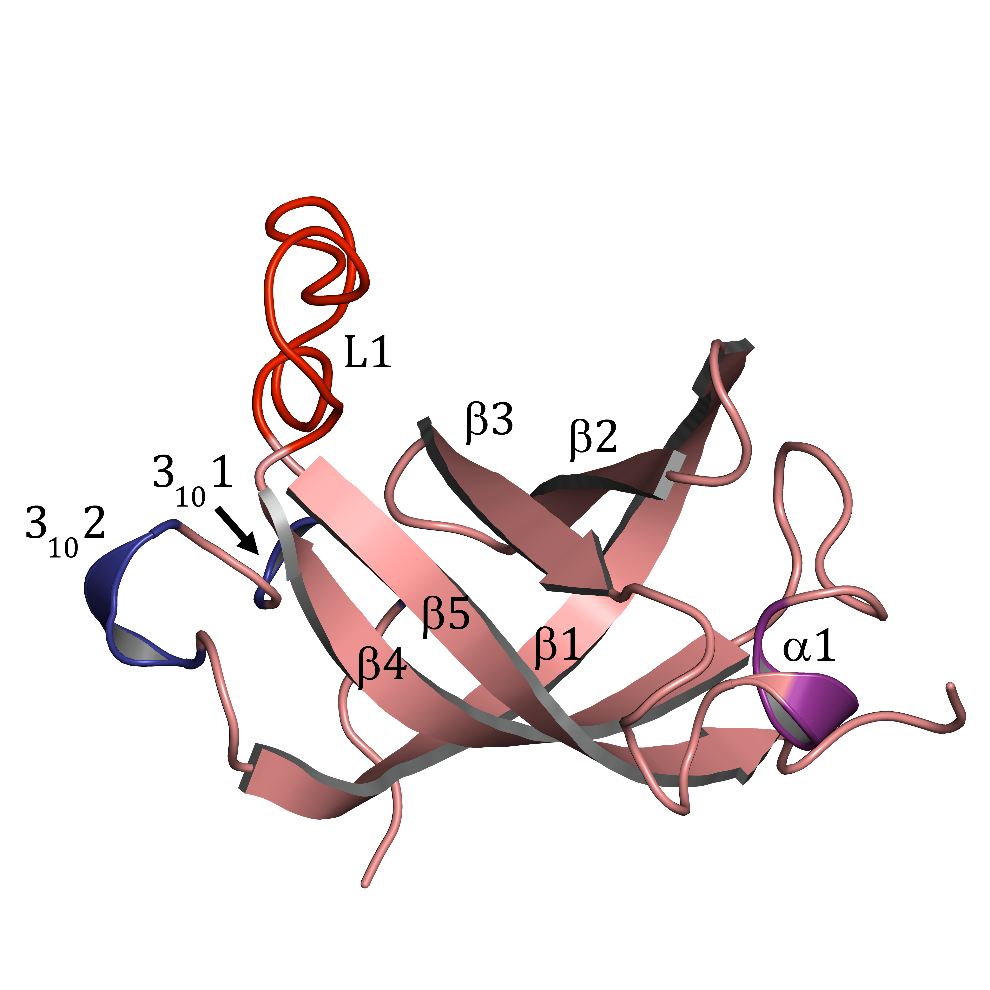
จากความสำคัญของเอนไซม์แอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสที่มีต่อกระบวนการสังเคราะห์โปรตีนในเซลล์สิ่งมีชีวิต ผศ. ดร. พิทักษ์ เชื้อวงศ์ อาจารย์ประจำภาควิชาเคมี คณะวิทยาศาสตร์ มหาวิทยาลัยเกษตรศาสตร์ บางเขน จึงสนใจศึกษาคุณสมบัติและโครงสร้างโปรตีนแอสปาร์ทิลทีอาร์เอ็นเอซินทิเทส ส่วนที่จับกับแอนติโคดอนของทีอาร์เอ็นเอ โปรตีนแอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสที่ศึกษาเป็นโปรตีนจากแบคทีเรีย Helicobacter pylori ซึ่งเป็นแบคทีเรียแกรมลบและก่อโรคในมนุษย์ การติดเชื้ออาจทำให้เกิดโรคแผลในกระเพาะอาหาร ซึ่งนำไปสู่โรคมะเร็งกระเพาะอาหารได้ ดังนั้นการทำความเข้าใจในโครงสร้างของเอนไซม์ดังกล่าวจึงเป็นข้อมูลพื้นฐานที่สำคัญ
คณะวิจัยได้ผลิตโปรตีนบริสุทธิ์ ผลการวิเคราะห์โดยเทคนิค Dynamic light scattering พบว่าโปรตีนมีความเสถียรในช่วงอุณหภูมิที่ทดสอบระหว่าง 20 ถึง 42 องศาเซลเซียส คณะวิจัยสนใจศึกษาโครงสร้างของเอนไซม์ดังกล่าวเพื่อนำไปสู่ความเข้าใจในความจำเพาะต่อทีอาร์เอ็นเอ เทคนิค Circular Dichroism Spectroscopy (CD) ให้ข้อมูลโครงสร้างแบบแผ่นพับซ้อนซึ่งมีความเสถียรที่อุณหภูมิสูง ซึ่งสอดคล้องกับผลการทดลองจากเทคนิค Dynamic Light Scattering (DLS)
ในการวิเคราะห์โครงสร้างสามมิติของเอนไซม์ ด้วยเทคนิค Protein X-ray Crystallography ดร. ชมภูนุช ส่งสิริฤทธิกุล นักวิทยาศาสตร์ระบบลำเลียงแสงของสถาบันวิจัยแสงซินโครตรอนได้เตรียมผลึกของโปรตีนแอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสในสารละลายที่ประกอบด้วยแอมโมเนียมซัลเฟต บัฟเฟอร์ซิเทรต และโพลีเอธิลีนไกลคอล 4,000 ในอัตราส่วนความเข้มข้นที่เหมาะสม (รูปที่ 1A; อุณหภูมิบ่ม 18 องศาเซลเซียส เป็นเวลา 2 วัน ด้วยเทคนิค Microbatch และ 1B; อุณหภูมิบ่ม 4 องศาเซลเซียส เป็นเวลา 6 วัน ด้วยเทคนิค Vapor diffusion) ซึ่งสามารถเก็บข้อมูลการเลี้ยวเบนรังสีเอกซ์ได้ความละเอียดระดับ 2.0 อังสตรอม มี unit-cell parameters a = b = 61.83, c = 141.38 Å จัดอยู่ในกลุ่มสมมาตรแบบ tetragonal และมี space group แบบ P41212 โดยหนึ่ง asymmetric unit ประกอบด้วยโปรตีนสองโมเลกุล และมีน้ำเป็นองค์ประกอบ 57.83%

รูปที่ 1 ผลึกของโปรตีนแอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสส่วนที่จับกับแอนติโคดอนของทีอาร์เอ็นเอ จากแบคทีเรีย H. Pylori 1A) เตรียมได้ในเบื้องต้นด้วยเทคนิค Microbatch 1B) เตรียมได้โดยปรับสภาวะการทดลองจาก 1A) ด้วยเทคนิค Vapor diffusion
โครงสร้างโปรตีนมีลักษณะเป็น Oligosaccharide-Binding (OB) fold (รูปที่ 2) เมื่อเปรียบเทียบโครงสร้างสามมิติของเอนไซม์จากแบคทีเรีย H. Pylori กับแบคทีเรียอื่นๆ พบว่ามีความคล้ายทั้งรูปร่างและขนาด (รูปที่ 3) แต่มีโครงสร้าง turn ที่แตกต่างกัน ซึ่งส่งผลให้เอนไซม์จากแบคทีเรีย H. Pylori มีโครงสร้างที่ compact มากกว่า นอกจากนี้ด้วยคุณสมบัติของ turn E และ turn F ทำให้เอนไซม์สามารถจับได้ทั้งแอนติโคดอนของทีอาร์เอ็นเอแอสพาติก แอซิด tRNAAsp (34GUC36) และ ทีอาร์เอ็นเอแอสพาราจีน tRNAAsn (34GUU36) ซึ่งโครงสร้างที่วิเคราะห์ได้บ่งชี้ว่ากรดอะมิโนโพรลีนตำแหน่งที่ 84 มีความสำคัญที่น่าจะมีผลต่อความจำเพาะของเอนไซม์
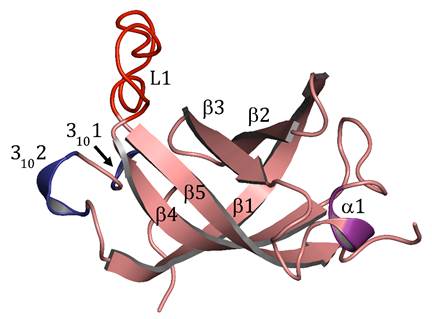
รูปที่ 2 โครงสร้างสามมิติของเอนไซม์แอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสส่วนที่จับกับแอนติโคดอนของทีอาร์เอ็นเอ จากแบคทีเรีย H. Pylori (PDB code 5GRO)
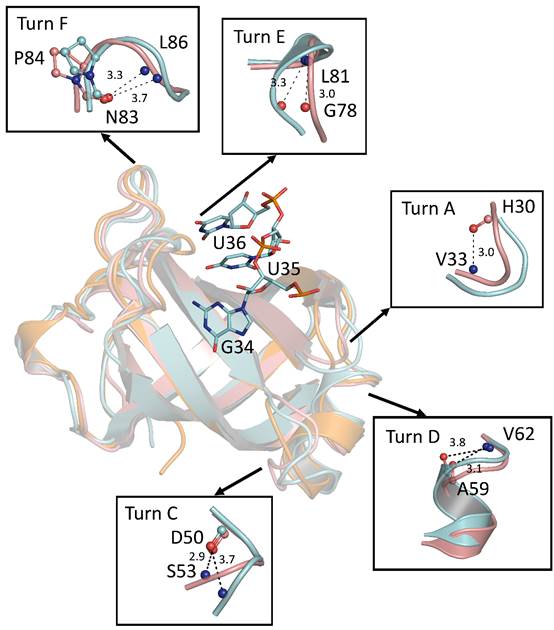
รูปที่ 3 โครงสร้างสามมิติของเอนไซม์แอสปาร์ทิลทีอาร์เอ็นเอซินทิเทสส่วนที่จับกับแอนติโคดอนของทีอาร์เอ็นเอ จากแบคทีเรีย H. Pylori (PDB code 5GRO; สีชมพูส้ม) เปรียบเทียบกับโครงสร้างจากแบคทีเรีย Pseudomonas aeruginosa (PDB code 4WJ4; สีฟ้าเขียว) และจากแบคทีเรีย Mycobacterium smegmatis (PDB code 4O2D; สีส้ม)
คณะวิจัยโครงการฯ
- พิชญดา เฟื่องฟูลอย, สุวิมล สืบค้า และ ผศ. ดร. พิทักษ์ เชื้อวงศ์ (ภาควิชาเคมี คณะวิทยาศาสตร์ มหาวิทยาลัยเกษตรศาสตร์ บางเขน)
- ดร. ชมภูนุช ส่งสิริฤทธิกุล (ระบบลำเลียงแสงที่ 7.2W: Macromolecular Crystallography (MX) สถาบันวิจัยแสงซินโครตรอน)
- Prof. Dr. Chun-Jung Chen (Scientific Research Division, National Synchrotron Radiation Research Center, Hsinchu, Taiwan)
เอกสารอ้างอิง
- Cathopoulis, T., Chuawong, P. & Hendrickson, T. L. (2007). Novel tRNA aminoacylation mechanisms. Molecular Biosystem. 3: 408–418.
- Chuawong, P. & Hendrickson, T. L. (2006). The nondiscriminating aspartyl-tRNA synthetase from Helicobacter pylori: anticodon-binding domain mutations that impact tRNA specificity and heterologous toxicity. Biochemistry. 45: 8079-8087.
- Fuengfuloy, P., Chuawong, P., Suebka, S., Wattana-amorn, P., Williams, C., Crump, P. M. & Songsiriritthigul, (2013). Overproduction of the N-terminal anticodon-binding domain of the non-discriminating aspartyl-tRNA synthetase from Helicobacter pylori for crystallization and NMR Measurements. Protein Expression and Purification. 89(1): 25-32. DOI: 10.1016/j.pep.2013.02.006.
- Songsiriritthigul, C., Suebka, S., Chen, C-J., Fuengfuloy, P. & Chuawong, P. (2017) Crystal structure of the N-terminal anticodon-binding domain of non-discriminating aspartyl-tRNA synthetase from Helicobacter pylori. Acta Crystallographica F: Structural Biology Communications. F73, 62-69.




